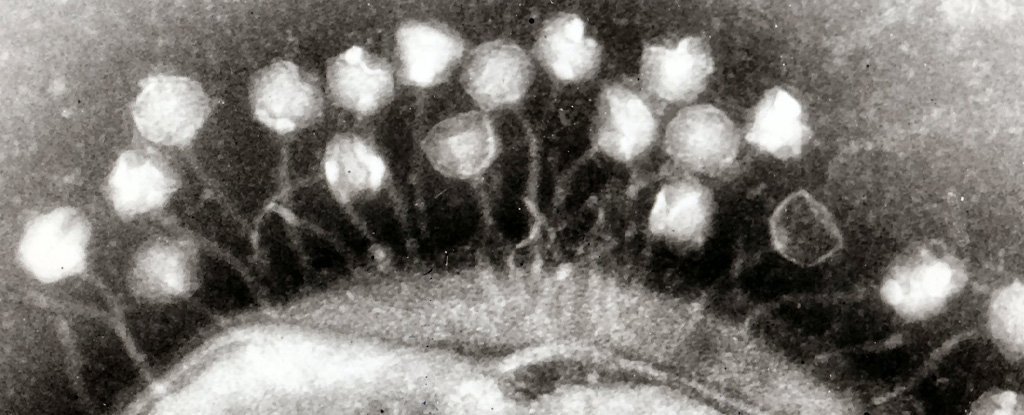
Em um estudo publicado hoje na Nature Microbiology, os pesquisadores identificaram 54.118 espécies de vírus que vivem no estômago humano, com 92 por cento deles previamente desconhecidos.
No entanto, como nós e nossos colaboradores do Joint Genome Institute e da Stanford University, na Califórnia, descobrimos, a grande maioria deles eram bacteriófagos, ou “fagos”. Esses vírus “comem” bactérias e são incapazes de infectar células humanas.
Quando a maioria de nós pensa em vírus, pensamos em organismos que causam doenças como caxumba, sarampo ou, mais recentemente, COVID-19. No entanto, os corpos humanos contêm um grande número desses minúsculos parasitas que se alimentam dos germes que ali residem.
Todo mundo defeca (mas nem todo cocô é igual)
Recentemente, houve um surto de interesse no microbioma intestinal humano: o grupo de bactérias que vivem em nosso trato gastrointestinal.
Além de nos ajudar a digerir nossos alimentos, esses microrganismos desempenham uma variedade de outras atividades críticas. Eles nos protegem de microrganismos perigosos, regulam nossa saúde mental, preparam nosso sistema imunológico quando jovens e continuam a desempenhar um papel no controle imunológico durante a vida adulta.
É seguro afirmar que o intestino humano é agora a ecologia microbiana mais pesquisada do mundo. Apesar disso, mais de 70% das espécies microbianas que ali vivem não foram cultivadas em laboratório.
Sabemos disso porque uma técnica chamada metagenômica nos permite acessar os projetos genéticos do microbioma intestinal. Esta é uma abordagem altamente eficaz em que o DNA é coletado diretamente do ambiente e sequenciado aleatoriamente, fornecendo-nos um instantâneo do que está presente e o que pode estar fazendo.
A pesquisa metagenômica demonstrou o quão longe ainda precisamos ir para catalogar e isolar todas as espécies microbianas encontradas no intestino humano – e ainda mais no caso de vírus.
11.810 amostras de fezes
Nós e nossos colegas extraímos computacionalmente sequências virais de 11.810 metagenomas fecais disponíveis publicamente de indivíduos em 24 países diferentes para nossa nova pesquisa. Procuramos determinar até que ponto os vírus colonizaram o intestino humano.
Esse esforço resultou na criação do catálogo Metagenomic Gut Virus, o recurso mais abrangente de seu tipo até o momento. Esta coleção contém 189.680 genomas virais que representam mais de 50.000 espécies virais únicas.
Surpreendentemente (ou talvez inesperadamente), mais de 90% dessas espécies virais são desconhecidas para a ciência. Eles codificam em conjunto mais de 450.000 proteínas únicas, representando um vasto reservatório de potencial funcional que pode ser útil ou destrutivo para seus hospedeiros microbianos e, em última análise, humanos.
Além disso, examinamos subespécies de vários vírus e descobrimos que vários exibiam padrões regionais notáveis nas 24 nações pesquisadas.
Por exemplo, uma subespécie de crAssfago recentemente caracterizada e intrigante era abundante na Ásia, mas era rara ou inexistente em amostras da Europa e da América do Norte. Isso pode ser resultado da disseminação restrita do vírus em populações humanas específicas.
Durante nossa viagem de campo molecular, uma das funções mais frequentemente descobertas foi a dos retroelementos geradores de diversidade (DGRs). São uma classe de elementos genéticos que alteram certos genes-alvo para gerar variações favoráveis ao hospedeiro. Isso pode ajudar os vírus em sua corrida armamentista evolutiva contínua com seus hospedeiros bacterianos no caso dos DGRs.
Surpreendentemente, descobrimos que um terço das proteínas virais codificadas com mais frequência têm atividades desconhecidas, incluindo mais de 11.000 genes associados a “beta-lactamases” que conferem resistência a antibióticos como a penicilina.
Estabelecendo uma ligação entre os vírus intestinais e suas bactérias hospedeiras
Depois de identificar os fagos, precisamos conectá-los a seus hospedeiros microbianos. Os CRISPRs, mais reconhecidos por suas inúmeras aplicações na edição de genes, são sistemas imunológicos bacterianos que “lembram” infecções virais anteriores e trabalham para prevenir sua recorrência.
Eles conseguem isso copiando e armazenando bits do vírus invasor em seus próprios genomas, que podem então ser utilizados para direcionar e eliminar o vírus especificamente em encontros futuros.
Usamos esse registro histórico de ataques para conectar um grande número de genomas virais a seus hospedeiros no ecossistema intestinal. Sem surpresa, espécies virais muito abundantes foram associadas a espécies bacterianas altamente abundantes no intestino, a maioria das quais eram membros dos filos Firmicutes e Bacteroidota.
Então, o que devemos fazer com todos esses dados novos? O tratamento com fagos é uma aplicação promissora de um banco de dados de vírus intestinais e seus hospedeiros. A terapia fágica é uma noção antiga que antecede os antibióticos, nos quais os vírus são empregados para tratar doenças visando especificamente patógenos bacterianos.
Tem havido um debate sobre a possibilidade de adaptar os microbiomas intestinais das pessoas para melhorar sua saúde por meio de mudanças na dieta, probióticos, prebióticos ou mesmo “transpoosões” (transplantes de microbiota fecal).
A terapia fágica pode contribuir para esse objetivo, aumentando a precisão da manipulação do microbioma em nível de espécie ou mesmo de subespécie. Por exemplo, o patógeno bacteriano Clostridioides difficile (ou Cdiff, para abreviar) é uma das principais causas de diarreia hospitalar e pode ser alvejado principalmente por fagos.
O tratamento com fagos pode permitir uma modulação mais matizada de populações de bactérias não patogênicas no intestino. Um compêndio abrangente de vírus intestinais é um excelente ponto de partida para buscar tais objetivos aplicáveis.
Vale a pena enfatizar, entretanto, que as estimativas baseadas em nossos achados indicam que examinamos apenas uma porcentagem da diversidade viral intestinal total. Como resultado, ainda temos um longo caminho a percorrer.
Philip Hugenholtz, Professor de Microbiologia da Escola de Química e Biociências Moleculares da University of Queensland, e Soo Jen Low, Pesquisador de Pós-Doutorado da University of Queensland.
Este artigo foi republicado de The Conversation sob uma licença Creative Commons. Leia o artigo original em inglês.